IT之家 9 月 25 日消息,科技媒体 9to5Mac 昨日(9 月 24 日)发布博文,报道称苹果研究团队发布了轻量级蛋白质折叠预测 AI 模型 SimpleFold,采用流匹配(Flow Matching)方法替代多序列比对(MSA)等复杂模块,大幅降低计算成本。
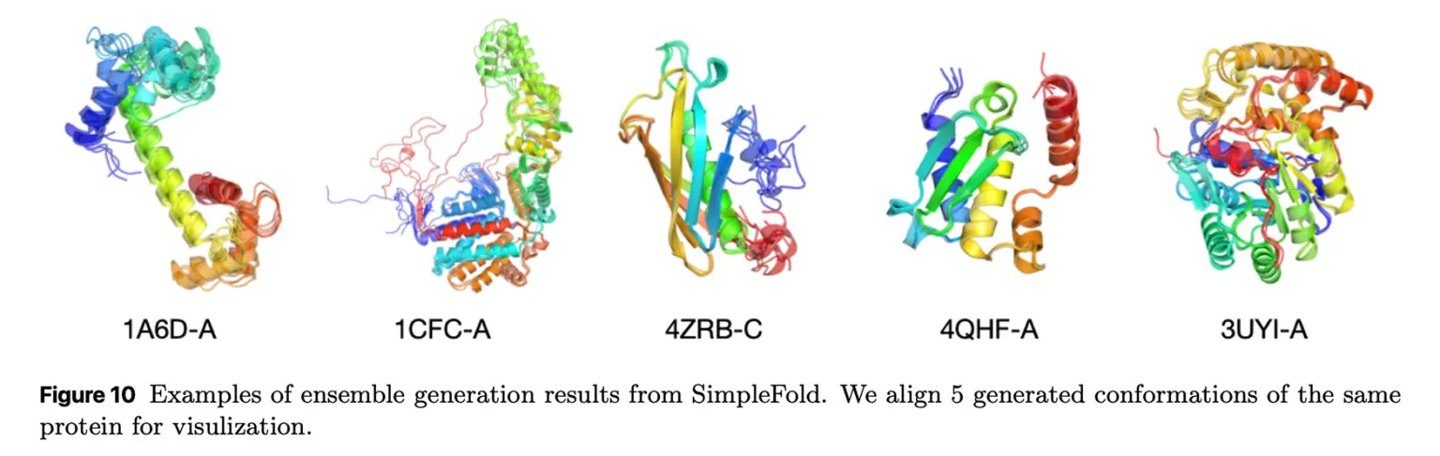
该媒体指出 AlphaFold2(Google DeepMind 的突破性 AI 模型)、RoseTTAFold 等传统模型虽然具备更高的精度,但由于依赖多序列比对、三角更新等计算密集的特定架构,训练与推理成本高昂。苹果团队希望通过简化结构,降低硬件门槛,让更多科研团队受益。
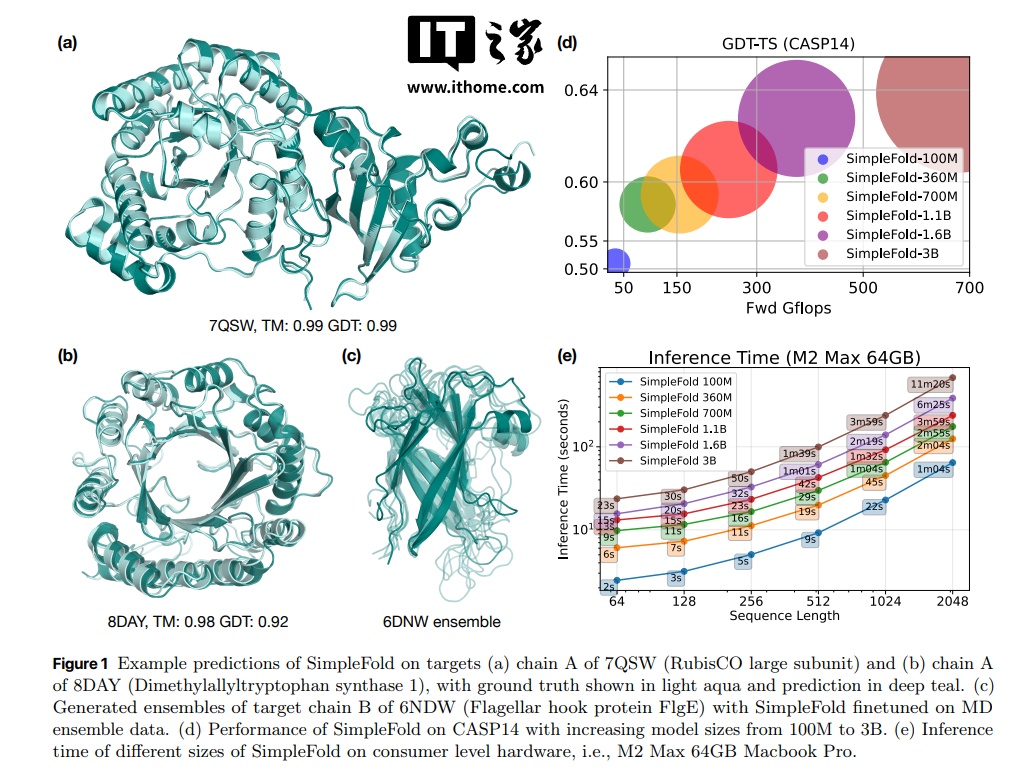
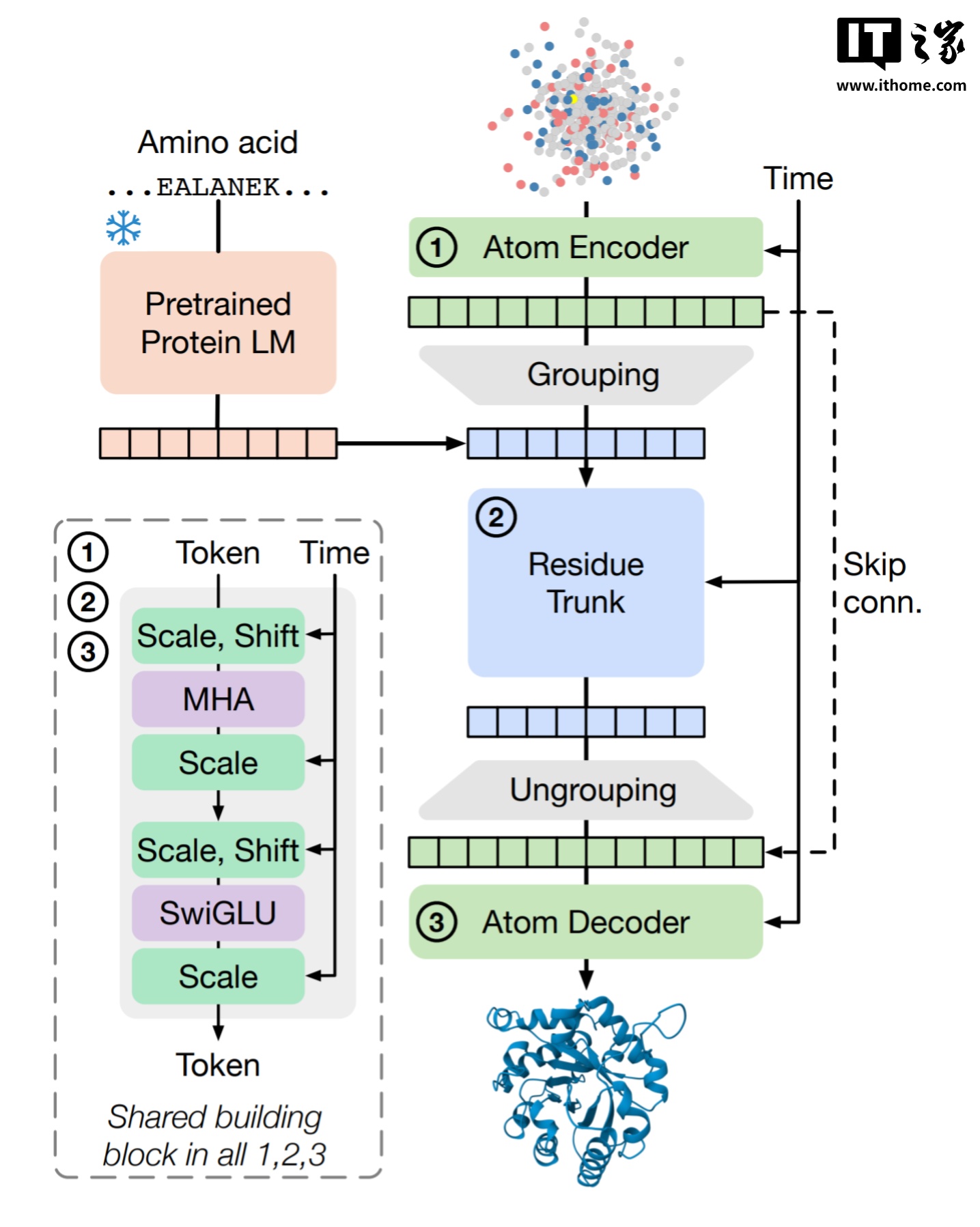
SimpleFold 核心采用 2023 年提出的流匹配(Flow Matching)方法,这种技术源自扩散模型,但在生成过程中不逐步去噪,而是直接从随机噪声一次生成目标结构。相比逐步迭代的扩散模型,这种方式跳过多个去噪环节,计算量更低、生成速度更快,已在文本生成图像和 3D 建模领域获得成功。
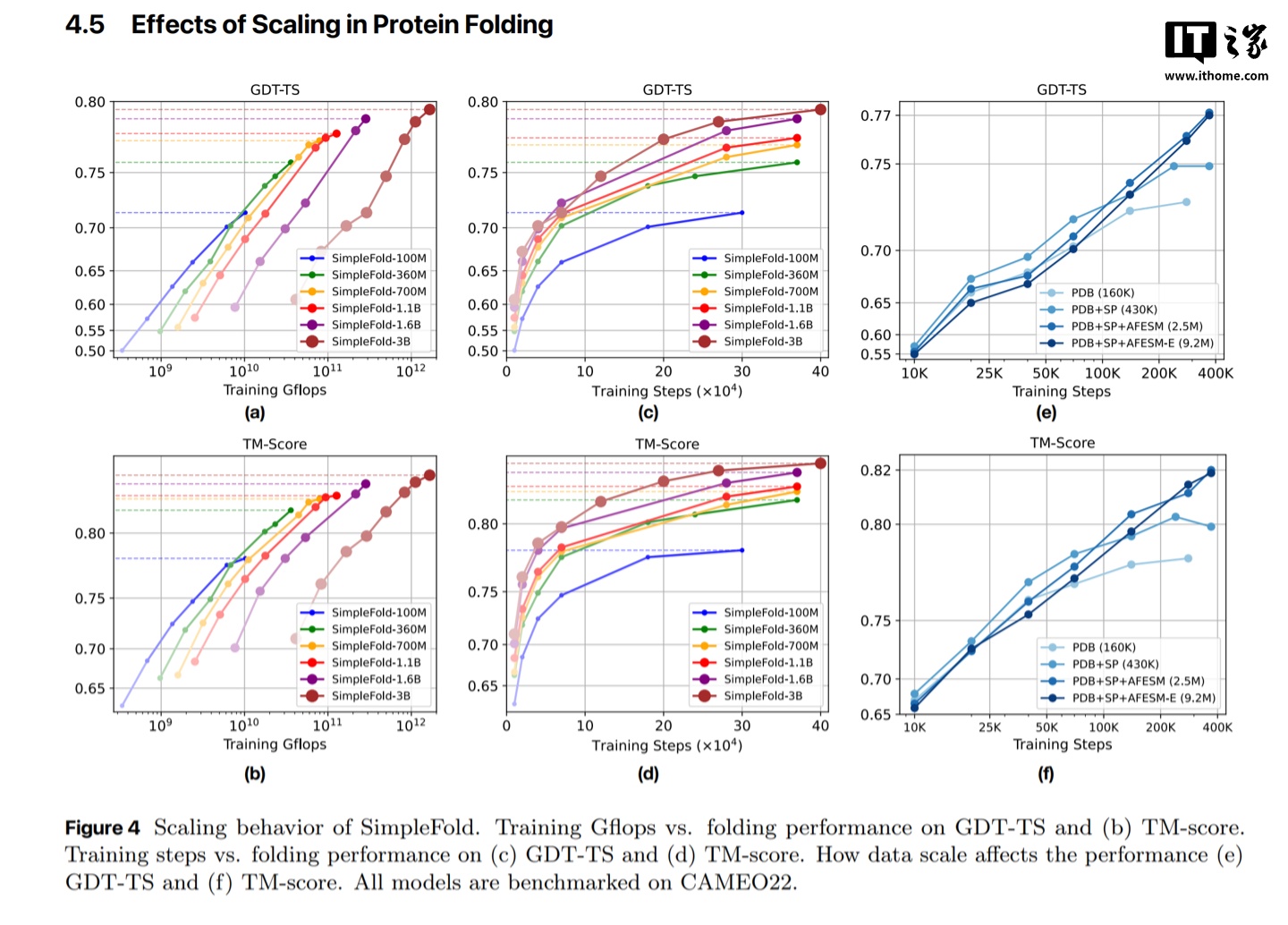
在实验中,研究人员分别训练了从 100M 到 3B 参数的多个版本,并在 CAMEO22 和 CASP14 两大权威基准上进行评估。
这些测试覆盖了模型在泛化性、鲁棒性(Robustness,一个系统或过程在面临干扰、不确定性或异常输入时,仍能维持其功能或稳定运行的能力)和原子级精度上的表现,结果显示,SimpleFold 在性能上稳定优于同类流匹配模型 ESMFlow,并媲美顶尖模型表现。
具体来看,SimpleFold 无需使用昂贵的多序列比对和三角注意机制,在 CAMEO22 上达到 AlphaFold2、RoseTTAFold2 约 95% 的性能。更小的 SimpleFold-100M 版本在保持高效的同时,性能超过 ESMFold 的 90%,验证了通用架构模块在蛋白质预测领域的可行性和竞争力。
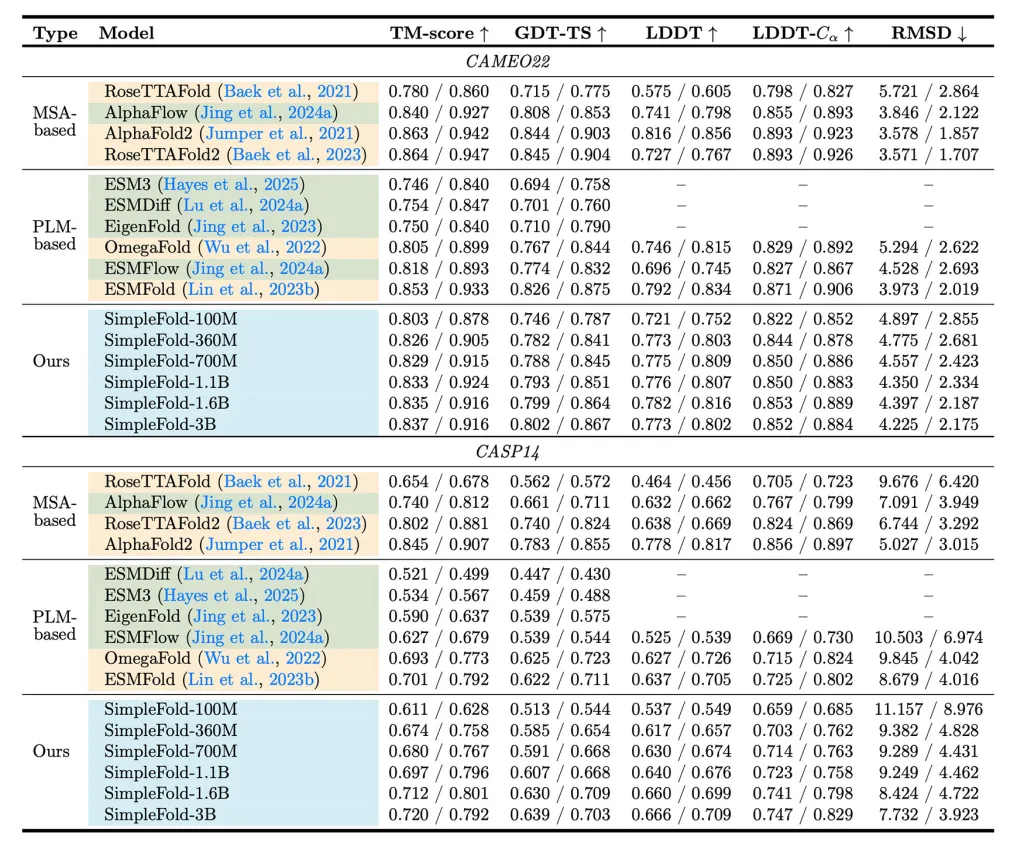
研究还发现,随着模型规模和训练数据增加,性能稳定提升,尤其在高难度测试中优势明显。苹果团队强调,SimpleFold 只是起点,希望它能推动社区构建更多高效、强大的蛋白质生成模型,进一步加速药物研发和新材料探索。
IT之家附上参考
Apple researchers develop SimpleFold, a lightweight AI for protein folding prediction
SimpleFold: Folding Proteins is Simpler than You Think



